10. Juli 2023
LIONESS: Mit Lasern Gewebe darstellen
Umfangreiche Zusammenarbeit am ISTA ermöglicht einzigartigen „Live“- Einblick in Komplexität des Gehirns
In einer neuen Publikation, die heute in der Fachzeitschrift Nature Methods veröffentlicht wurde, hat nun ein interdisziplinäres Team von Wissenschafter:innen des Institute of Science and Technology Austria (ISTA) eine neuartige Methode zur Beobachtung der Struktur und Dynamik des Gehirns vorgestellt.

Das menschliche Gehirn ist eines der kompliziertesten Organe, mit denen sich Wissenschafter:innen je befasst haben. Vollgepackt mit einer fast unermesslichen Menge an Informationen, ist das Gehirn mit seinem Netzwerk aus rund 86 Milliarden Neuronen das höchstentwickelte Computersystem. Eine solche Komplexität zu verstehen ist äußerst schwierig und bedarf Technologien, mit denen die winzigen, komplizierten Wechselwirkungen, die im Gehirn auf mikroskopischer Ebene stattfinden, entschlüsselt werden können. Bildgebende Verfahren sind daher entscheidende Instrumente in den Neurowissenschaften.
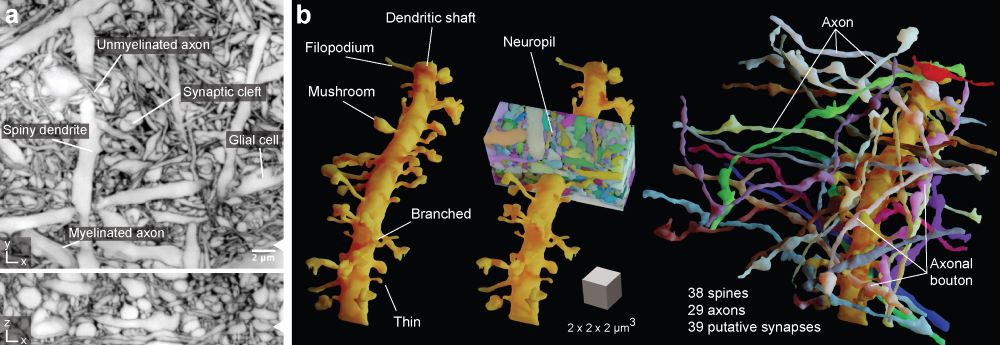
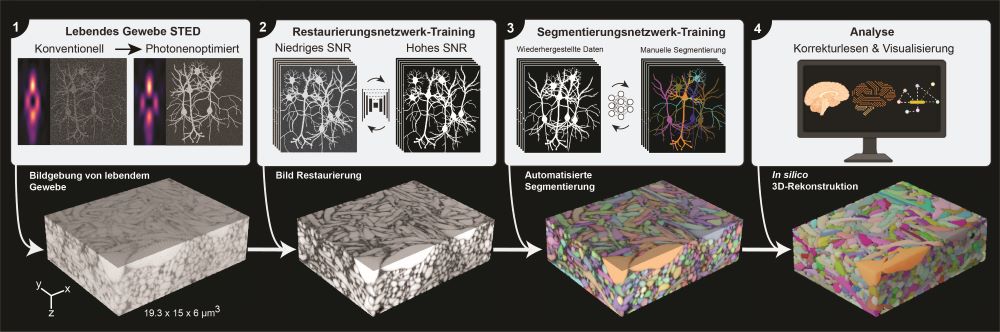
Die von Johann Danzls Gruppe am ISTA entwickelte neue Bildgebungs- und virtuelle Rekonstruktionstechnologie, ist ein großer Fortschritt in diesem Bereich und trägt den treffenden Namen LIONESS (Live Information Optimized Nanoscopy Enabling Saturated Segmentation). LIONESS ist eine Pipeline zur Abbildung, Rekonstruktion und Analyse von lebendem Hirngewebe mit einer bisher nicht dagewesen räumlichen Auflösung und Vollständigkeit.
„LIONESS ermöglicht uns zum ersten Mal, lebendes Hirngewebe umfassend und dicht zu rekonstruieren. Durch das mehrfache Abbilden des Gewebes, können wir mit LIONESS beobachten und messen, wie die dynamische Zellbiologie im Gehirn ihren Lauf nimmt“, so Erstautor Philipp Velicky. „Das Ergebnis ist ein dreidimensionales, rekonstruiertes Bild der zellulären Strukturen. Die vierte Dimension stellt die Zeit dar – die Probe kann nämlich über Minuten, Stunden oder Tage hinweg abgebildet werden“, fügt er hinzu.
Mit LIONESS können Neurowissenschafter:innen lebendes Hirngewebe untersuchen und hochauflösende 3D-Bilder erzeugen, ohne die lebende Probe zu beschädigen.
Die Stärken von LIONESS sind vor allem die verfeinerte Optik und die zwei Ebenen des Deep Learning, die den Kern des Systems bilden: Die erste Ebene verbessert die Bildqualität, während die zweite die verschiedenen zellulären Strukturen in der dichten neuronalen Umgebung identifiziert.
Die Pipeline ist das Ergebnis einer engen Zusammenarbeit zwischen verschiedenen Forschungsgruppen am Institute. Dazu zählen sowohl die Gruppe von Johann Danzl, die Gruppe von Bernd Bickel, die Gruppe von Peter Jonas und Gaia Novarinos Gruppe sowie die wissenschaftlichen Services (Scientific Service Units, SSUs) und internationale Kooperationspartner:innen. Danzl über die Zusammenarbeit: „„Wir haben eine dynamische Gruppe von Wissenschafter:innen mit einzigartiger Disziplinen-übergreifender Expertise zusammengestellt. Gemeinsam arbeiten wir daran, eine Technologielücke in der Analyse von Hirngewebe zu schließen.“
Hürden überwinden
Dank der Elektronenmikroskopie, die die untersuchende Probe auf der Grundlage ihrer Wechselwirkungen mit Elektronen abbildet, ist es möglich, Rekonstruktionen von Gehirngewebe zu erstellen. Mit dieser Technik kann man Bilder mit einer Auflösung von wenigen Nanometern – einem Millionstel Millimeter – aufnehmen, jedoch hat sie aber auch große Nachteile. Die Probe muss in einem biologischen Zustand fixiert und dann physisch geschnitten werden , um 3D-Informationen zu erhalten. Dynamischen Informationen können so nicht gewonnen werden.
Nicht physisch, sondern optisch wird intaktes Gewebe bei der Lichtmikroskopie geschnitten. Das Auflösungsvermögen dieser spezifischen Technik wird jedoch durch die Eigenschaften der Lichtwellen, die sie zur Erzeugung eines Bildes verwendet, stark eingeschränkt. Im besten Fall liegt die Auflösung bei einigen hundert Nanometern – viel zu grobkörnig, um wichtige zelluläre Details im Gehirngewebe zu erfassen.
Ansätze mit Super-resolution Light Microscopy (aus dem Englischen: superauflösende Lichtmikroskopie) durchbrechen diese Auflösungsgrenze. Die jüngsten Arbeiten in diesem Feld sind vielversprechend. „SUSHI“ (Super-resolution Shadow Imaging) beispielsweise kombiniert das Einfärben der Zellzwischenräume mit Farbstoffmolekülen mit der Nobelpreis-ausgezeichneten Superresolution-Technik STED (Stimulated Emission Depletion). Dadurch können superaufgelöste „Schatten“ der zellulären Strukturen offenbart werden und können so im Gewebe sichtbar gemacht werden. Trotzdem war es bisher nicht möglich, ganze Gehirnvolumina mit einer verbesserten Auflösung, die der komplexen 3D-Architektur des Gehirngewebes entspricht, abzubilden. Der Hauptgrund dafür ist, dass die höhere Auflösung auch eine hohe Lichtbelastung der Probe mit sich bringt, was dazu führt, dass das lebende Gewebe beschädigt wird oder gar „verschmort“.
Darin liegt genau die Stärke von LIONESS. Laut den Autor:innen versichert die Technik „schnelle und milde“ Bedingungen, sodass die untersuchte Probe am Leben bleibt. Dabei bietet die Methode eine isotrope Superauflösung, das heißt, sie ist in allen drei Raumdimensionen gleich gut und ermöglicht so die Visualisierung der zellulären Komponenten des Gewebes bis ins kleinste Detail (3D-Details im Nanomaßstab).
LIONESS sammelt während des Bildgebungsschritts nur so viele Informationen von der Probe, wie nötig. Danach folgt dann der erste Deep-Learning-Schritt, der über einen Prozess namens Image Restoration zusätzliche Informationen über die Struktur des Hirngewebes ergibt. Auf diese innovative Weise wird eine Auflösung von rund 130 Nanometern erreicht und ist dabei aber gleichzeitig schonend genug, um lebendes Hirngewebe in Echtzeit abzubilden. In weiterer Folge ermöglicht es einen zweiten Deep-Learning-Schritt, der die extrem komplexen Bilddaten interpretiert und die neuronalen Strukturen auf automatisierte Weise identifiziert.
Zielanflug
„Der interdisziplinäre Ansatz ermöglichte es uns, die miteinander verknüpften Beschränkungen des Auflösungsvermögens und der Lichtexposition des lebenden Systems zu überwinden. Außerdem können wir nun komplexe 3D-Daten sinnvoll nutzen und die zelluläre Architektur des Gewebes mit molekularen und funktionellen Messungen verknüpfen“, so Danzl.
Für die virtuelle Rekonstruktion arbeiteten Danzl und Velicky mit Expert:innen für visuelle Datenverarbeitung zusammen, darunter auch die Bickel Gruppe am ISTA und die Forschungsgruppe von Hanspeter Pfister an der Harvard Universität. Beide steuerten ihr Fachwissen im Bereich der automatischen Segmentierung – dem Prozess der automatischen Erkennung der Zellstrukturen im Gewebe – und der Visualisierung bei und wurden dabei von Christoph Sommer, Staff Scientist in der Imaging & Optics Facility am ISTA unterstützt. An den komplexen Markierungsstrategien waren Neurowissenschafter:innen und Chemiker:innen aus Edinburgh, Berlin und vom ISTA beteiligt. Dies ermöglichte, funktionelle Messungen zu überbrücken, d.h. die zellulären Strukturen zusammen mit der biologischen Signalaktivität im selben lebenden neuronalen Schaltkreis auszulesen. Dazu wurden in Zusammenarbeit mit der Jonas-Gruppe am ISTA die Kalziumionenflüsse in die Zellen abgebildet und die zelluläre elektrische Aktivität gemessen. Die Gruppe von Gaia Novarino steuerte menschliche Hirnorganoide bei – sogenannte Mini-Gehirne, mit denen man menschliche Gehirnentwicklung nachahmen kann. Zusätzlich wurde die Arbeit durch die fachliche Unterstützung der erstklassigen wissenschaftlichen Services des ISTA unterstützt.
Gehirnstruktur und -aktivität sind äußerst dynamisch. Die Strukturen entwickeln sich weiter, wenn das Gehirn neue Aufgaben ausführt und erlernt. Dieser Aspekt wird oft als „Plastizität“ bezeichnet. Die Veränderungen in der Gewebestruktur und dessen Beobachtung ist also von entscheidender Bedeutung, um die Geheimnisse hinter der Plastizität des Gehirns zu entschlüsseln. Das am ISTA entwickelte neue Verfahren bietet das Potenzial, subzelluläre Strukturen und deren Veränderungen im Laufe der Zeit aufzudecken – ein großer Schritt, um die funktionelle Architektur von Hirngewebe und anderen Organen zu verstehen.
Publikation:
Philip Velicky et al. 2023. Dense 4D nanoscale reconstruction of living brain tissue. Nature Methods. DOI:10.1038/s41592-023-01936-6
Projektförderung:
Der ISTA-Teil des Projektes wurde finanziell unterstützt durch Austrian Science Fund (FWF) (Grant Nos. I3600-B27, DK W1232, und Z 312-B27); Gesellschaft für Forschungsförderung NÖ (NFB) (Grant No. LSC18-022); ISTA Interdisciplinary project grant; EU Horizon 2020, Marie-Skłodowska Curie (Grant No. 665385), Marie Skłodowska-Curie Actions Individual Fellowship (101026635), European Research Council (Grant No. 715767 – Materializable, 715508 – Reverseautism, und 692692 – Giantsyn; und dem Human Frontier Science Program postdoctoral fellowship (LT000557/2018).



